Khi học Lý thuyết thì ta sẽ thấy các bài toán có vài đặc trưng (features) và vector input thường chỉ có độ dài 1,2 phần tử. Nhưng khi làm việc thực tế thì chúng ta sẽ thường xuyên phải deal với các input có số đặc trưng lớn, dài dằng dặc và chúng ta chả biết bỏ cái nào, dùng cái nào cho vừa hiệu quả vừa đỡ được chi phí tính toán.
Đó là lúc chúng ta nghĩ đến PCA để giảm chiều dữ liệu mà vẫn giữ lại được các đặc trưng tốt để phục vụ cho bài toán của chúng ta!
Nguồn baifi: Mì AI
Vậy PCA là gì?
Đọc thêm Principle Component Analysis (PCA) – nhập môn TIN & HỌC TiiL (tinhoc123.edu.vn)
PCA là viết tắt của Principal Component Analysis. Ta dịch thô sang tiếng Việt là “Phân tích thành phần chính”, tạm hiểu theo cách “nông dân” của mình là ta sẽ phân tích dữ liệu và sau đó tìm ra các thành phần chính của dữ liệu để giữ lại các thành phần đó. Ví dụ dữ liệu của chính ta có N features thì sau khi áp dụng PCA sẽ còn K features chính mà thôi (K<N).
Việc làm như trên sẽ giúp cho chúng ta:
- Giảm chiều dữ liệu mà vẫn giữ được đặc trưng chính, chỉ mất đi “chút ít” đặc trưng.
- Tiết kiệm thời gian, chi phí tính toán
- Dễ dàng visualize dữ liệu hơn để giúp ta có cái nhìn trực quan hơn.
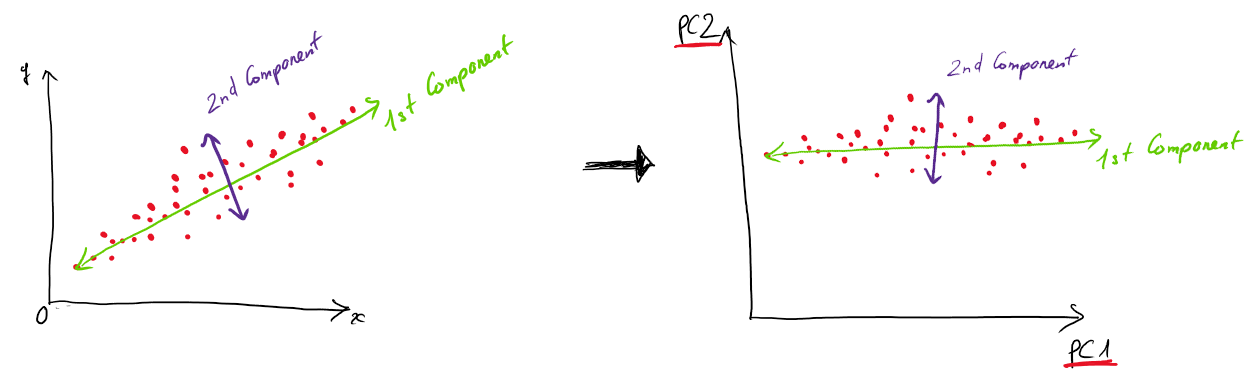
Các components ở đây ta nói thực chất là các vectors độc lập tuyến tính được chọn sao cho khi chiếu các điểm dữ liệu lên vector đó thì các điểm dữ liệu có sự variance lớn nhất ( biến động nhiều nhất, phương sai lớn nhất).

như hình trên, chúng ta chọn 2 vector component theo thứ tự: 1st Comp sẽ có mức độ variance lớn nhất, ta chọn trước, sau đó đến 2nd Comp…. và cứ thế. Khi làm thực tế chúng ta sẽ cần xác định hoặc thử sai xem sẽ chọn bao nhiêu components là hợp lý và mang lại kết quả tốt nhất.
Xét một cách nhìn khác thì PCA cũng là một bài toán chuyển hệ tọa độ như hình dưới:
Tại sao phải chọn comp với mức độ dữ liệu biến thiên variance lớn nhất làm gì?
Vì:
Ví dụ xét bài toán phân loại classification, ví dụ : Ung thư/ Không ung thư, Spam/Normal…. Bây giờ nếu chúng ta chọn 1 comp mà chiếu lên đó các điểm dữ liệu không high variance, nó đè lên nhau và co cụm lại một chỗ thì làm sao mà phân loại nổi. Nói cách khác là làm sao tìm được đường hay mặt phẳng chia tách các dữ liệu đó thành 2 phần khác nhau cho 2 class khác nhau. Do đó, ta phải chọn comp sao cho khi chiếu data lên comp đó thì nó high variance.
Phần 2 – Triển khai Principal Component Analysis với Python
Để demo cách chúng ta triển khai PCA với Python, mình sẽ dùng một bộ dữ liệu khá phổ biến và tích hợp sẵn trong Sklearn đó là Breast_Cancer – Ung thư vú. Dữ liệu này có rất nhiều features khác nhau và khá lằng nhằng (do mình không có kiến thức y khoa haha) và dùng nó để minh họa PCA là chuẩn bài rồi.
Đầu tiên là cứ phải import đầy đủ các thư viện cái đã. Phần này là thói quen của mình khi làm việc với data, mình cứ import hết đề phòng thiếu
import pandas as pd import numpy as np import matplotlib.pyplot as plt import seaborn as sns %matplotlib inline
Okie, xong rồi! Bây giờ ta sẽ load dữ liệu in ra xem data của chúng ta như nào:
from sklearn.datasets import load_breast_cancer # Đọc dữ liệu từ sklearn cancer_set = load_breast_cancer() # Chuyển thành DataFrame cancer_data = pd.DataFrame(data=cancer_set ['data'], columns=cancer_set['feature_names']) cancer_data.head()
Và ta thấy dữ liệu có cả mớ cột 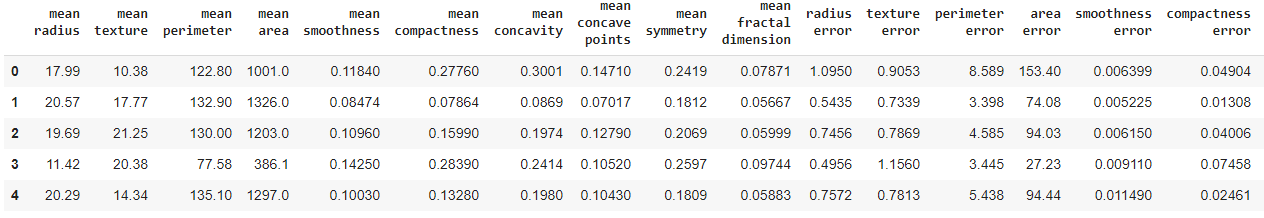
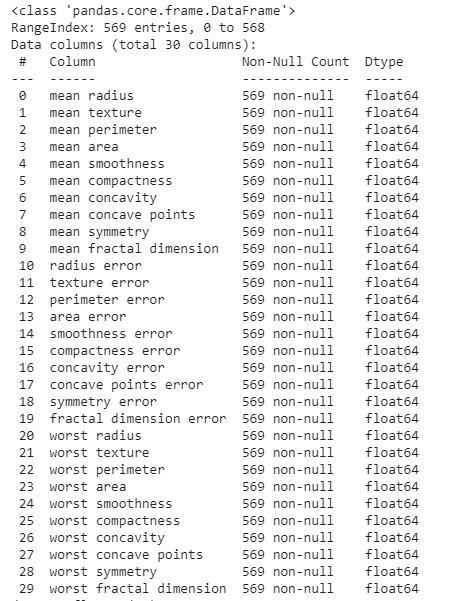
Nhìn qua dữ liệu chúng ta thấy range của mỗi cột khá lệch nhau, ta áp dụng chuẩn hóa nào:
from sklearn.preprocessing import StandardScaler scaler = StandardScaler() # Fit vào dữ liệu scaler.fit(cancer_data) # Thực hiện transform scale scale_cancer_data = scaler.transform(cancer_data)
Sau bước này ta sẽ làm việc với scale_cancer_data nhé!
Rồi bây giờ thực hiện PCA:
from sklearn.decomposition import PCA # Khởi tạo đối tượng PCA với số comp = 2 my_pca = PCA (n_components = 2 ) # Fit vào data my_pca.fit(scale_cancer_data) # Thực hiện transform pca_scale_cancer_data = my_pca.transform(scale_cancer_data)
Bây giờ chúng ta sẽ kiểm tra xem có thực sự dữ liệu của chúng ta đã giảm chiều không bằng cách in ra 2 món này:
print("Dữ liệu gốc: ", scale_cancer_data.shape)
# Dữ liệu gốc: (569, 30)
print("Dữ liệu sau PCA:" , pca_scale_cancer_data.shape)
# Dữ liệu sau PCA: (569, 2)
Ngon rồi nhé, đã chỉ còn 2 chiều.
Bây giờ mình sẽ show cho các bạn thấy rằng khi chọn 2 chiều có variance lớn nhất thì chúng ta sẽ dễ dàng thực hiện phân tách các lớp trong bài toán classification như nào nha.
Chúng tá sẽ plot đống này lên đồ thị:
plt.figure(figsize = (10,6)) # Thành phần comp số 1 pca_1 = pca_scale_cancer_data[:, 0] # Thành phần comp số 2 pca_2 = pca_scale_cancer_data[:, 1] # Vẽ đồ thị plt.scatter(x=pca_1, y = pca_2)
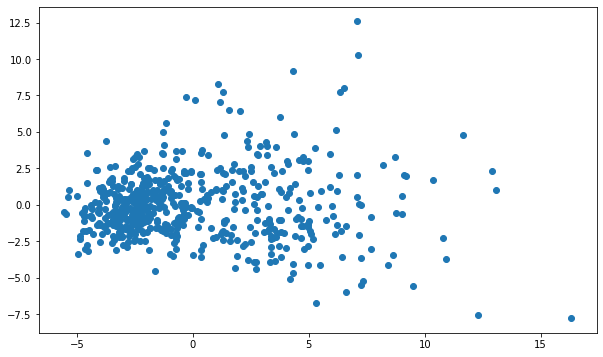
Để ý rằng trong dữ liệu đầu vào của chúng ta có phần target – là nhãn của dữ liệu (1 là ung thư và 0 là ngược lại). Ta in ra thử xem:
Rồi, bây giờ ta sửa lại code plot tý chút, thêm cái mầu sắc của từng điểm dữ liệu theo class của nó xem sao:
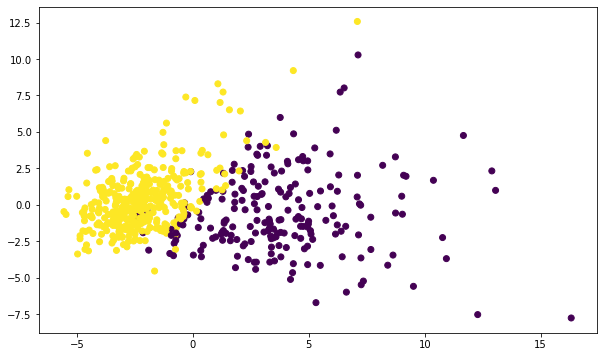
plt.figure(figsize = (10,6)) # Thành phần comp số 1 pca_1 = pca_scale_cancer_data[:, 0] # Thành phần comp số 2 pca_2 = pca_scale_cancer_data[:, 1] # Vẽ đồ thị plt.scatter(x=pca_1, y = pca_2, c = cancer_set['target'])
Awsome, các điểm dữ liệu giữa ung thư và không ung thư đã được phân biệt khá rõ ràng, chúng ta thấy có thể dễ dàng vẽ được một đường thẳng (hoặc siêu phẳng) phân chia 2 class này như trong hình dưới:
Chúng ta sẽ đi nốt phần cuối, vậy thì sau khi giảm chiều từ hơn 30 về 2 thì thực sự cái pca_scale_cancer_data là gì? In ra phát nào:
my_pca.components_
array([[ 0.21890244, 0.10372458, 0.22753729, 0.22099499, 0.14258969,
0.23928535, 0.25840048, 0.26085376, 0.13816696, 0.06436335,
0.20597878, 0.01742803, 0.21132592, 0.20286964, 0.01453145,
0.17039345, 0.15358979, 0.1834174 , 0.04249842, 0.10256832,
0.22799663, 0.10446933, 0.23663968, 0.22487053, 0.12795256,
0.21009588, 0.22876753, 0.25088597, 0.12290456, 0.13178394],
[-0.23385713, -0.05970609, -0.21518136, -0.23107671, 0.18611302,
0.15189161, 0.06016536, -0.0347675 , 0.19034877, 0.36657547,
-0.10555215, 0.08997968, -0.08945723, -0.15229263, 0.20443045,
0.2327159 , 0.19720728, 0.13032156, 0.183848 , 0.28009203,
-0.21986638, -0.0454673 , -0.19987843, -0.21935186, 0.17230435,
0.14359317, 0.09796411, -0.00825724, 0.14188335, 0.27533947]])
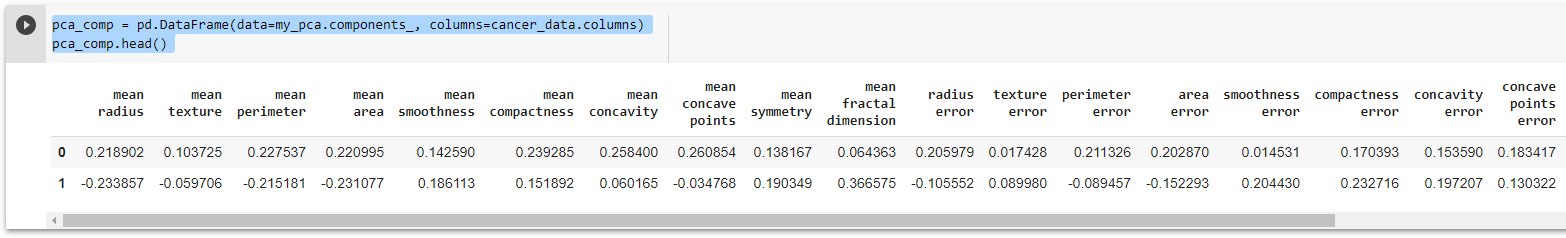
Để ý bạn sẽ thấy nó có số dòng = số component và số cột = số features gốc. Để làm rõ hơn ta chuyển nó thành dataframe:
pca_comp = pd.DataFrame(data=my_pca.components_, columns=cancer_data.columns) pca_comp.head()
Và ta quan sát một chút sẽ thấy 2 ông comp, mỗi ông 1 dòng và mối tương quan với các features gốc của dữ liệu (phép chiếu mà):